Capítulo 10 Otimização de Configurações Amostrais
10.1 Introdução
10.2 Estimativa de tendências
# Otimização para estimativa de tendências espaciais ####
# Cronograma de recozimento simulado
schedule <- spsann::scheduleSPSANN(
chains = 1000, initial.temperature = 50, stopping = 100,
x.max = 1540, y.max = 2060, x.min = 0, y.min = 0, cellsize = 40)
# Covariáveis espaciais:
# - soil: tipo de solo (3 categorias)
# - ffreq: frequência de inundação (3 categorias)
covars <- c("soil", "ffreq")
image(meuse.grid, "soil", axes = TRUE, col = terrain.colors(3))
image(meuse.grid, "ffreq", axes = TRUE, col = heat.colors(3))
# Otimização: somente no espaço de atributos
set.seed(2001)
res_dist_att <- spsann::optimDIST(
points = 15, candi = meuse.grid@coords, covars = meuse.grid@data[, covars],
schedule = schedule, plotit = TRUE)
# Otimização: em ambos os espaços de atributos e geográfico
set.seed(2001)
res_dist_geo <- spsann::optimDIST(
points = 15, candi = meuse.grid@coords, covars = meuse.grid@data[, covars],
use.coords = TRUE, schedule = schedule, plotit = TRUE)
# Comparar visualmente ambas as configurações #####
# Salvar imagem no formato PNG
dev.off()
png("images/optim_dist.png", height = 480 * 2, width = 480 * 3, res = 150)
par(mfrow = c(1, 2))
# Legenda
leg <- c("Sem coordenadas", "Com coordenadas")
# Tipo de solo
image(meuse.grid, "soil", axes = TRUE, col = terrain.colors(3))
title(main = "Tipo de solo", xlab = "Longitude (m)", ylab = "Latitude (m)")
points(res_dist_att$points[, 2:3], pch = 21)
points(res_dist_geo$points[, 2:3], pch = 24)
legend("topleft", pch = c(21, 24), legend = leg, bty = "n", title = "Covariáveis")
# Frequência de inundação
image(meuse.grid, "ffreq", axes = TRUE, col = cm.colors(3))
title(main = "Frequência de inundação", xlab = "Longitude (m)", ylab = "Latitude (m)")
points(res_dist_att$points[, 2:3], pch = 21)
points(res_dist_geo$points[, 2:3], pch = 24)
legend("topleft", pch = c(21, 24), legend = leg, bty = "n", title = "Covariáveis")
dev.off()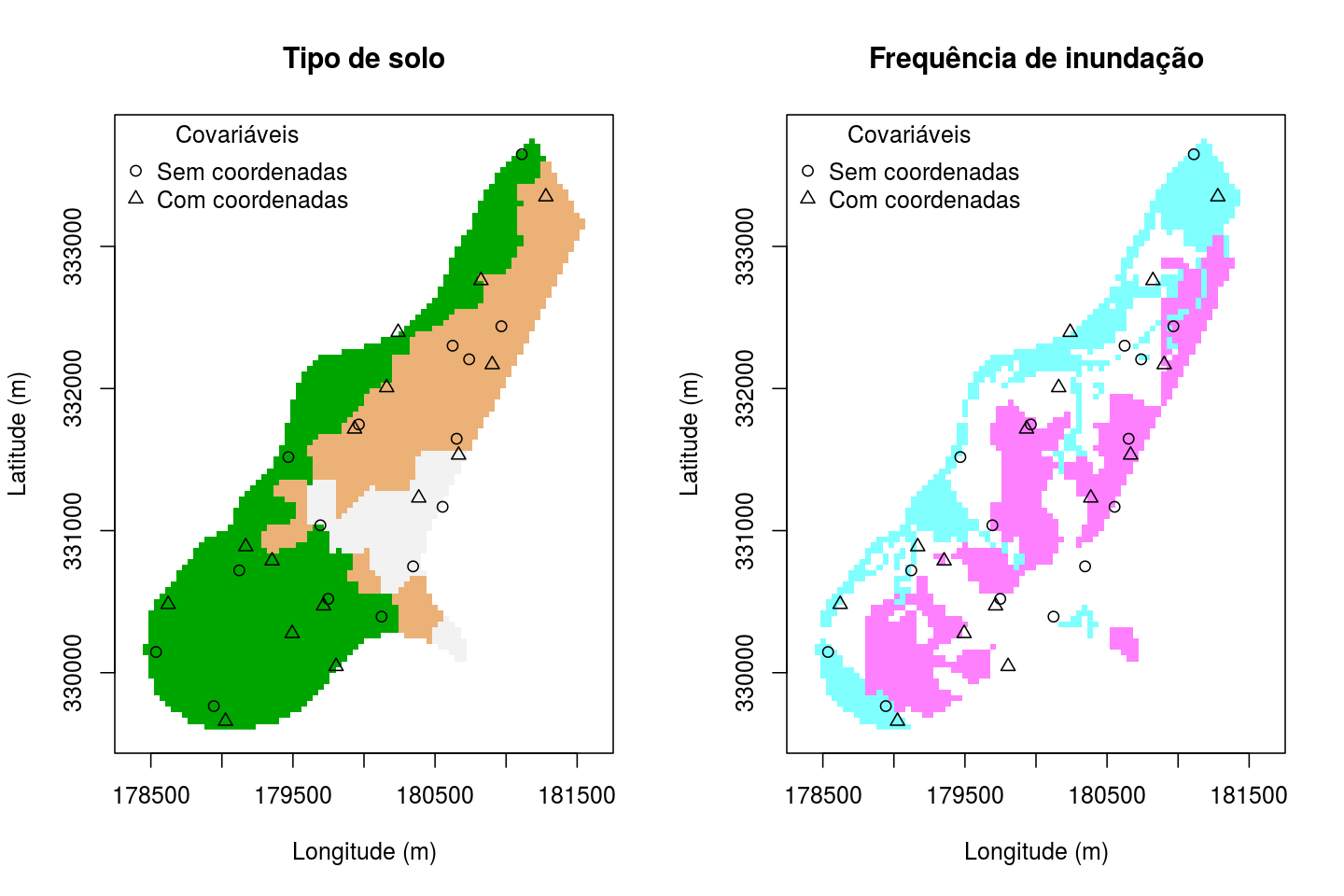
Configurações amostrais espaciais otimizadas no espaço de atributos (covariáveis) para estimativa detendências espaciais e o efeito de levar em consideração também o espaço geográfico.
10.3 Estimativa de variogramas
# Otimização para estimativa de variogramas ####
# Cronograma de recozimento simulado
schedule <- spsann::scheduleSPSANN(
chains = 1000, initial.temperature = 200, stopping = 100,
x.max = 1540, y.max = 2060, x.min = 0, y.min = 0, cellsize = 40)
# Otimização: pontos por classe de distância
set.seed(1984)
res_ppl_points <- spsann::optimPPL(
points = 15, candi = meuse.grid@coords, schedule = schedule, plotit = TRUE)
# Otimização: pares de pontos por classe de distãncia
set.seed(1984)
res_ppl_pairs <- spsann::optimPPL(
points = 15, candi = meuse.grid@coords, schedule = schedule, plotit = TRUE, pairs = TRUE)
# Comparar visualmente ambas as configurações #####
# Salvar imagem no formato PNG
dev.off()
png("images/optim_ppl.png", width = 480 * 3, height = 480 * 2, res = 150)
par(mfrow = c(1, 2))
# Pontos
image(meuse.grid, "soil", axes = TRUE, col = terrain.colors(3))
title(xlab = "Longitude (m)", ylab = "Latitude (m)", main = "Pontos por classe de distância")
points(res_ppl_points$points[, 2:3], pch = 21)
text(meuse.grid@bbox[1, 1], meuse.grid@bbox[2, 2], labels = "(a)")
# Pares de pontos
image(meuse.grid, "soil", axes = TRUE, col = terrain.colors(3))
title(xlab = "Longitude (m)", ylab = "Latitude (m)", main = "Pares por classe de distância")
points(res_ppl_pairs$points[, 2:3], pch = 21)
text(meuse.grid@bbox[1, 1], meuse.grid@bbox[2, 2], labels = "(b)")
dev.off()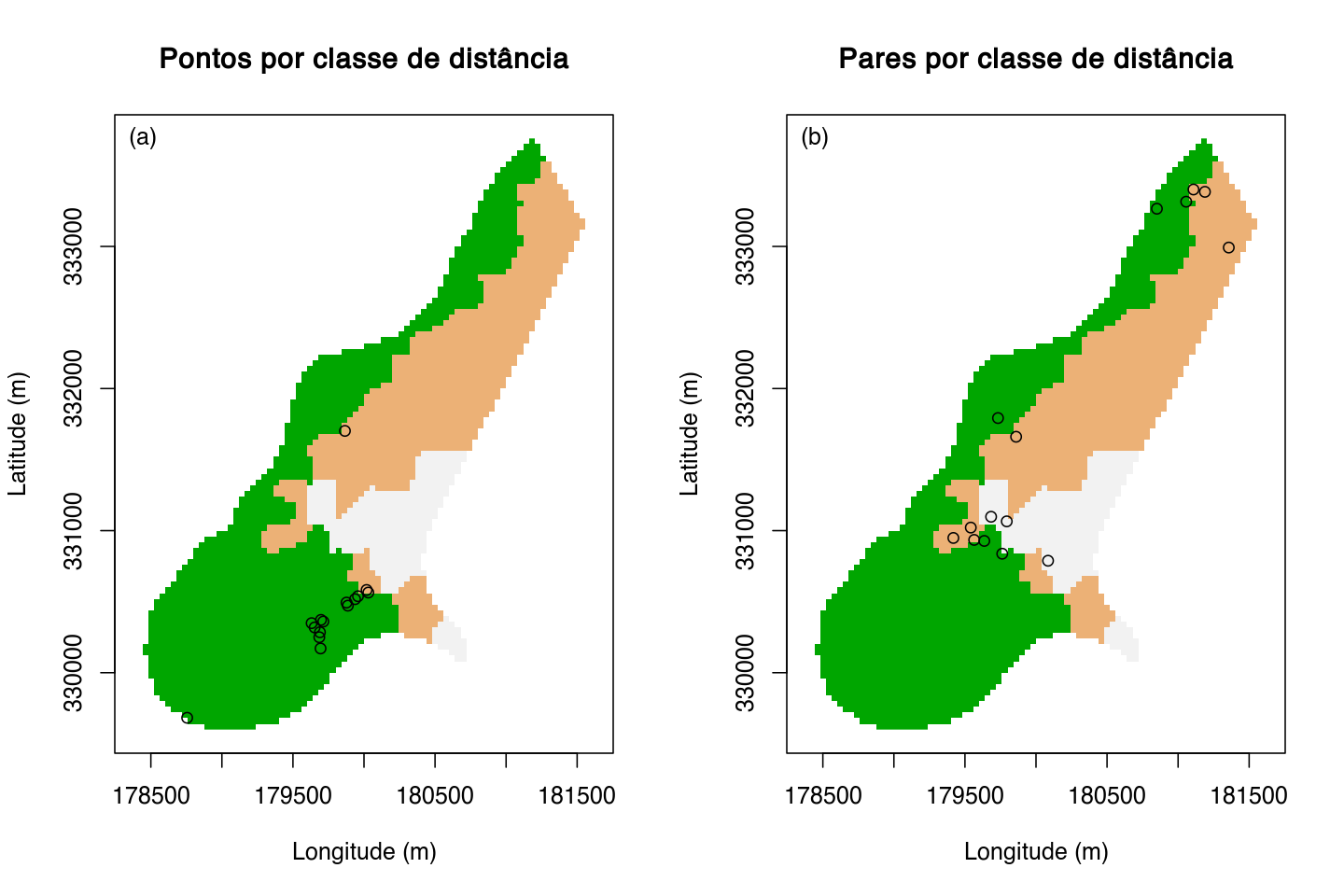
Configurações amostrais espaciais otimizadas para estimativa de variogramas usando como critério a distribuição do número de (a) pontos e (b) pares de pontos entre sete classes de distãncia, exponencialmente crescentes, entre pares de pontos.
10.4 Interpolação espacial
# Otimização para estimativa de tendências espaciais ####
# Cronograma de recozimento simulado
schedule <- spsann::scheduleSPSANN(
chains = 1000, initial.temperature = 500000, stopping = 100,
x.max = 1540, y.max = 2060, x.min = 0, y.min = 0, cellsize = 40)
# Otimização
set.seed(1984)
res_mssd <- spsann::optimMSSD(
points = 15, candi = meuse.grid@coords, schedule = schedule, plotit = TRUE)
# Salvar imagem no formato PNG
dev.off()
png("images/optim_mssd.png", width = 480 * 3, height = 480 * 2, res = 150)
# Tipo de solo
image(meuse.grid, "soil", axes = TRUE, col = terrain.colors(3))
title(xlab = "Longitude (m)", ylab = "Latitude (m)")
points(res_mssd$points[, 2:3], pch = 21)
dev.off()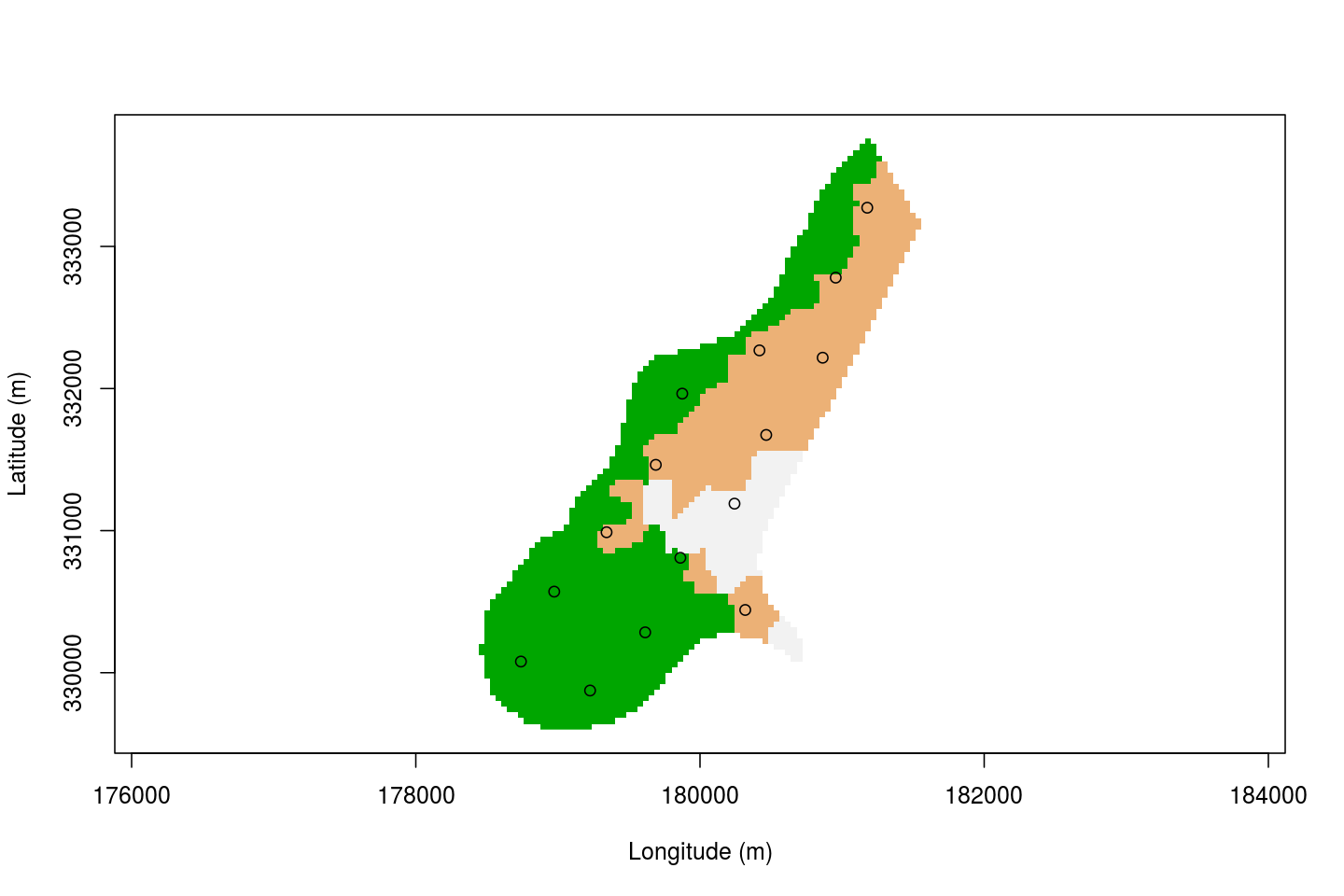
Configuração amostral espacial otimizada no espaço geográfico para fins de interpolação espacial usandokrigagem.